這裡將帶你從了解系統、安裝系統開始,再針對各種基因體分析需求,跟您說明如何使用基因體分析者系統來執行分析流程,我們的學習地圖做了三面向的規畫 , 內容包含了原理的介紹 , 實機的操作,及進一步資料解析的應用小工具 ,分門別類成以下三個系列:
 系列影片將從環境建置開始,帶您一步一步進入RNAseq、WES/WGS、microbiome的生資分析。
系列影片將從環境建置開始,帶您一步一步進入RNAseq、WES/WGS、microbiome的生資分析。 系列影片會介紹進入分析需要配備的基本武器,此外,亦提供能協助進階數據資料分析與圖形化的實用平台。
系列影片會介紹進入分析需要配備的基本武器,此外,亦提供能協助進階數據資料分析與圖形化的實用平台。 系列影片則由專家跟您分享及解說各種生資分析相關的原理及知識。
系列影片則由專家跟您分享及解說各種生資分析相關的原理及知識。
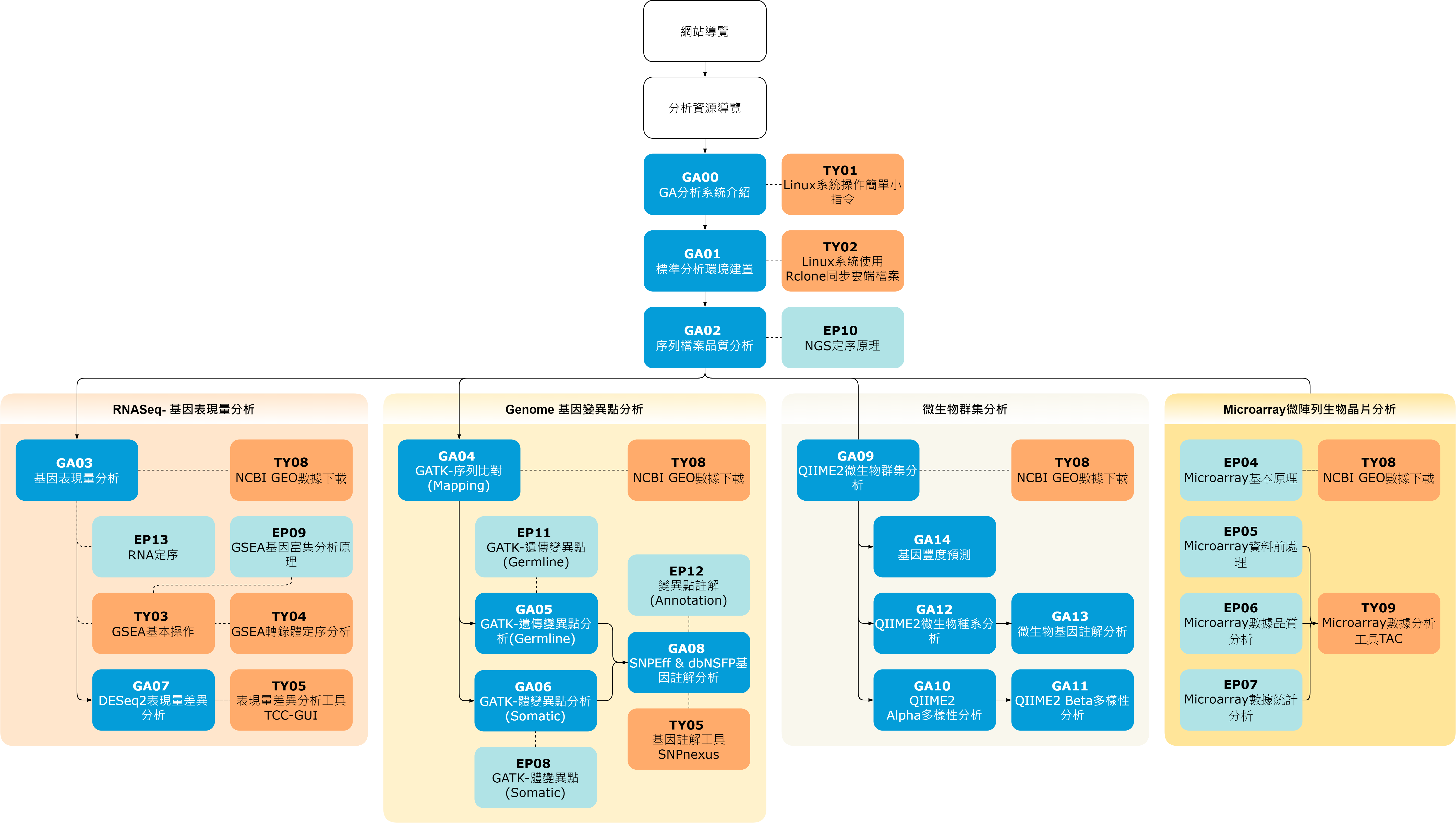
我們建議您先看完基因分析者系統介紹及Linux系統操作的相關指令,接著進行標準分析環境建置,以上部分完成, 便可著手序列資料品質分析,確認資料品質沒有問題後,就可以針對您有興趣的分析領域 (RNAseq_基因表現量分析、Genome_基因變異點分析、QIIME2_微生物群集分析),跟著學習地圖您也可以輕易走入生資分析的世界喔 ! 如果有問題, 可以留言在網頁或影片下方,我們會儘快回覆您。
 | 基本分析知識: 首先我們會介紹GA 系統的設計原理,讓大家了解如何使用我們的分析系統, 另外也會先讓大家了解一些基因體分析會用到的基礎技能, 這一部份包括在Linux系統中操作的指令 。 GA00 GA分析系統介紹 : 沒有資訊背景的生醫研究人員常常找不到進入生資領域的門路? 了解GA分析者系統是您開啟生資分析之路的鎖匙 , 讓我們一起踏上 生資分析之路吧! TY01 Linux系統操作簡單小指令: Genome Analyst(GA)分析使用的軟體是基於Linux系統開發, 這邊我們先來熟悉後續會常使用到的Linux相關指令。 |
 | GA標準分析環境 : GA系統使用 Conda 系統管理軟體 , 我們將其作成了一個安裝套件 (GApp) , 分析除了軟體系統環境外,還需要很多輔助的資源, 目前在國網中心台灣杉三號主機 (Taiwania III) 上,我們已完成這個資料集稱為Genome Analyst Bundle (GA Bundle)相關資料夾的建置, 提供給您直接使用 。 GA01 標準分析環境建置: 這裡將帶你申請國網中心的使用權限、 在台灣杉三號主機建置conda環境 , 以及取得GA_bundle使用權限並建置好GApp標準分析環境 , 準備好動手分析了嗎~~讓我們從建置分析環境開始吧! TY02 Linux系統使用Rclone同步雲端檔案:  GA中軟體的操作是使用Linux系統,分析資料的檔案相當龐大,我們會選擇存放在雲端,這集將向大家分享如何在Linux系統中使用Rclone 軟體同步雲端檔案。 |
 | GA02 序列檔案品質分析:  當你拿到定序下機後的RAW檔案 , 就可以跟著影片來進行分析,檢視這批序列檔案的品質情況囉~ EP10 NGS定序原理: 解析基因體或轉錄體最直接的方法就是把所有序列定量出來,在過去很難想像如何把這多不勝數的序列讀完, 次世代定序技術不但做到了這一點,還把成本降到一千美金,開啟了一個新的世代,快來看看他們是怎麼作到的。 |
 | RNAseq_基因表現量分析 : 透過次世代定序 (NGS) 對樣品中RNA做高通量定序,由於基因轉錄出來的RNA片段,與定序產出的read數成正比,因此基因 序列數量(read count),可用來代表該基因的表現量。 近年的RNAseq還包含了single cell sequencing及組織中的 in situ sequencing。 EP13 RNA定序 你知道RNA定序的原理嗎?在送定序之前需要注意什麼呢? 請收看本集的內容。 GA03 RNAseq_基因表現量分析:  當您完成序列檔案品質分析後 , 會得到一個將品質不好的reades清理過的.fastq檔案 , 這支影片將帶您將數百萬條的序列 跟reference genome進行比對 , 透過數個軟體的應用計算出每一個基因表現的reads 數 , 並將數據結果統整成一個excel檔案 , 方便您後續閱讀報告 。 GA07 DESeq表現量差異分析:  利用DESeq2軟體,幫助您快速在實驗中找到差異表現基因(Differential Expression Genes, DEG) 。 TY03 GSEA基本操作 : 基因組豐度分析(Gene set enrichment analysis) 是作基因體或轉錄體分析時常用的分析方法,這支影片將從軟體安裝開始, 用官方網站下載的Affymetrix 晶片資料,作一個簡單的操作示範 。 TY04 GSEA轉錄體定序分析 : 基因組豐度分析(Gene set enrichment analysis) 是作基因體或轉錄體分析時常用的分析方法,這支影片將由一個轉錄體定序分析 的結果 (RNA seq FPKM),建立 GCT 檔進行分析 。 TY05 表現量差異分析工具TCC-GUI:  利用軟體輕鬆繪製PCA,Heatmap及Volcano plot 等圖,得以協助我們快速找尋實驗中的差異 表現基因, 研究者可藉由基因差異表現在生物體中之意義進行實際的實驗探討。 TY08 NCBI GEO 數據下載:  |
 | Genome_基因變異點分析: 全基因體定序 (whole genome sequencing, WGS) 與全外顯子定序 (whole exome sequencing, WES) NGS的資料分析最複雜 也最重要的一個步驟就是變異點偵測(Variant Calling), GATK (Genome Analysis ToolKit)提出了有效的方法作變異位點偵測, 是目前最主流的基因變異點分析軟體之一,可以進行遺傳變異點分析及體變異分析,隨著不斷發展,目前也可以用於人類以外的 其他物種,亦可用於CNV (copy number variation)和SV ( structural variation)的檢測。 EP11 GATK-遺傳變異點(Germline): 分析變異位點直覺的就是把樣品的序列跟正常人的序列比對一下就好了,但事實上沒有這麼簡單,因為技術及生物的背景雜訊, 以前分析變異位點偵測錯誤率高的沒法使用,直到GATK 出來提出了有效的方法作變異位點偵測,來看看現在的變異位點偵測 是如何做的吧~~~ EP08 GATK-體突變點(Somatic):  GA04 GATK-序列比對(mapping):  GA05 GATK-遺傳變異點分析(Germline):  GA06 GATK-體變異點分析(Somatic):  GA08 SNPEff & dbNSFP基因註解析:  EP12 變異點註解(Annotation):  TY08 NCBI GEO 數據下載:  |
 | QIIME2_微生物群集分析: QIIME2是一個功能強大具擴展性的微生物數據分析軟體,主要包括時間和空間分析和可視化工具,使研究人員能從原始序列數據開始進行代謝組和基因組數據分析,輸出之數據統計結果,可用於擴展微生物組分析功能,進行豐度預測、多樣性分析及微生物基因註解,自動化數據來源追溯確保數據的可重複,微生物組數據透明化。 GA09 QIIME2 微生物群集分析:  GA14 基因豐度預測:  GA12 QIIME2 微生物種系分析 :  GA10 QIIME2 Alpha多樣性分析 :  GA13 微生物基因註解分析:  GA11 QIIME2 Beta多樣性分析 :  TY08 NCBI GEO 數據下載:  |
 | Microarray_微陣列生物晶片分析: Microarray 稱之生物晶片是一種微型化概念, 將預先合成好的數千或數萬個 核酸探針佈放於玻片載體上,當核酸探針與檢體的核酸 片段進行雜交反應時,可經由雷射掃描器顯示生物晶片的發光影像,透過核酸探針上螢光染劑的發光程度可得知特定基因的表現量,可同步且在短時間內提供大量基因序列資訊,有快速、精確、低成本之生物分析檢驗能力 。 EP04 Microarray基本原理: 生物晶片是作轉錄體分析很常用的工具,做一次實驗就能分析所有基因,威力強大! 讓我們來看看晶片的原理、平台及設計。 EP05 Microarray 資料前處理: 本集會討論Micorarray 訊號的量度、數據在分析前作如何處理及其中的理論基礎,學好這些概念進入實戰區才不會摸不著頭緒喔~ EP06 Microarray數據品質分析: 生物晶片數據品質分析是整個流程中非常重要的步驟,有好的品質把關,才會得到很棒的分析結果,快來看看怎麼作QC。 EP07 Microarray數據統計分析: 晶片上有千千萬萬的點,我們要用什麼統計方法才能挑出真得有改變的目標基因呢? 請跟著我們 收看本集的節目。 TY08 NCBI GEO 數據下載:  TY09 Microarray 數據分析工具TAC:  |